お知らせ
2022年12月28日 【プレスリリース】新発見!CRMP1遺伝子の変異がもたらす神経発達障害の機序を解明
新発見!CRMP1遺伝子の変異がもたらす神経発達障害の機序を解明
| Point ○ 東京女子医科大学・生化学分野の新敷信人助教、瀧澤光太郎助教、中村史雄教授の研究グループは、ドイツ・シャリテー・ベルリン医科大学、中国・広州医科大学との共同研究により、神経系に発現するCRMP1遺伝子のミスセンス変異が神経発達障害をもたらすことを新たに見出しました。 1.3名の神経発達障害の患者さんにCRMP1遺伝子のミスセンス変異を見出しました。 2.種を越えて高度に保存されたアミノ酸にミスセンス変異が生じていました。 3.ミスセンス変異はCRMP1タンパク質の多量体形成を阻害し、神経細胞の発達を抑制しました。 4.変異CRMP1の複合体形成を促進させて、神経発達障害を回復できる可能性があります。 |
Ⅰ 研究の背景と経緯
神経発達に関与する遺伝子の変異が様々な精神・神経疾患の原因となりうることが、マウス等のモデル生物の実験から示されています。しかしヒト神経発達に関わる疾患において特定の遺伝子の変異が示されたものはまだごく一部に限られます。私たちは今までに神経発達に関わる様々な分子の機能を培養神経細胞やノックアウトマウスを用いて明らかにしてきました。そのような分子の1つであるCollapsin Response Mediator Protein 1 (CRMP1)のチロシンリン酸化が、神経細胞の樹状突起形成に関わることを2021年に報告しています (Kawashima T et al, J Neurochem 2021)。今回、本学はドイツおよび中国の研究者と協力して、神経発達障害の患者さんにおいて見出されたCRMP1遺伝子の変異が、神経突起の伸長に影響を及ぼすことを明らかにしました。
Ⅱ 研究の内容
神経発達障害の患者さん3名の遺伝子解析から、神経発達に関わるCRMP1遺伝子の変異が同定されました。3名の患者さんは共に両親には変異が見出されない新規変異(de novo variant)であり、いずれもCRMP1翻訳領域のアミノ酸置換を伴うミスセンス変異であることがわかりました(図1)。
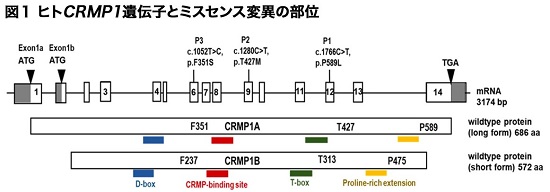
CRMP1は神経系に多く発現し、発達期の神経細胞において神経突起の伸長やシナプス形成に関わる分子です。またCRMP1のノックアウトマウスは記憶障害や統合失調症に関連する行動を示すことが報告されています。しかしヒト疾患におけるCRMP1のミスセンス変異と神経発達障害の関係は今まで報告されていませんでした。
3名の患者さんで見出されたCRMP1変異は、それぞれ異なるアミノ酸のミスセンス変異でした。いずれもCRMP1遺伝子において、種を越えて高度に保存されているアミノ酸残基の変異をもたらすものでした。
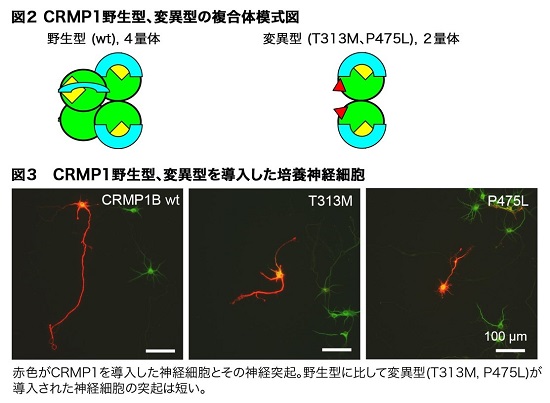
CRMP1タンパク質は4つが会合して1つの複合体を形成します(図2, 野生型)。今回見出されたCRMP1のミスセンス変異は、いずれもCRMP1タンパク質の複合体形成に影響を与えることが、タンパク質構造解析ソフトウエアを用いた検討から推測されました。そこで野生型と変異型のCRMP1を組換えタンパク質として調製し、ゲル濾過クロマトグラフィーを用いて複合体の大きさを測定しました。その結果、野生型に較べて変異型の複合体が小さくなることが示されました(図2, 変異型)。
さらに野生型、変異型のCRMP1をマウスの神経細胞に発現させたところ、変異型のCRMP1は神経突起の伸長を抑制することがわかりました(図3)。これらの結果から、神経発達障害の患者さんで見出されたCRMP1遺伝子のミスセンス変異はCRMP1タンパク質の多量体形成を阻害して、神経回路発達の遅延や障害をもたらすと考えられました。
3名の患者さんで見出されたCRMP1変異は、それぞれ異なるアミノ酸のミスセンス変異でした。いずれもCRMP1遺伝子において、種を越えて高度に保存されているアミノ酸残基の変異をもたらすものでした。
CRMP1タンパク質は4つが会合して1つの複合体を形成します(図2, 野生型)。今回見出されたCRMP1のミスセンス変異は、いずれもCRMP1タンパク質の複合体形成に影響を与えることが、タンパク質構造解析ソフトウエアを用いた検討から推測されました。そこで野生型と変異型のCRMP1を組換えタンパク質として調製し、ゲル濾過クロマトグラフィーを用いて複合体の大きさを測定しました。その結果、野生型に較べて変異型の複合体が小さくなることが示されました(図2, 変異型)。
さらに野生型、変異型のCRMP1をマウスの神経細胞に発現させたところ、変異型のCRMP1は神経突起の伸長を抑制することがわかりました(図3)。これらの結果から、神経発達障害の患者さんで見出されたCRMP1遺伝子のミスセンス変異はCRMP1タンパク質の多量体形成を阻害して、神経回路発達の遅延や障害をもたらすと考えられました。
Ⅲ 今後の展開
CRMP1の上流には神経回路形成を制御するセマフォリン3A (Sema3A)が位置します。またCRMP1はいくつかのリン酸化酵素によりリン酸化されて、神経突起伸長やシナプス形成などの機能を果たします。今後はCRMP1が担うSema3Aの情報伝達経路においてCRMP1のミスセンスが、リン酸化修飾も含めどのような影響を与えるかを検討していきます。さらに多量体形成によりCRMP1の機能が回復するのであれば、ミスセンス変異CRMP1の複合体形成を促す化合物の探索といった取り組みも今後検討していきます。
【お問い合わせ先】
<研究に関すること>
中村 史雄(ナカムラ フミオ)
東京女子医科大学 医学部生化学分野 教授
〒162-8666 東京都新宿区河田町8-1
Tel&Fax:03-3353-8112 (Ext: 31525) 直通 03-5269-7415
E-mail: nakamura.fumio@twmu.ac.jp
Angela M Kaindl, MD PhD, Professor
Department of Pediatric Neurology & Institute of Cell Biology and Neurobiology
CharitéCrossOver (CCO), Virchowweg 6
Charité - Universitätsmedizin Berlin
Berlin, Germany
Phone: +49-(0)30 450 528429
E-mail: angela.kaindl@charite.de
Hao Hu, PhD, Professor
Laboratory of Medical Systems Biology, Guangzhou Women and Children's Medical Center, Guangzhou Medical University
School of Medicine, South China University of Technology
9 Jinsui Road, Guangzhou, Guangdong, 510623, P.R.China
Phone: +86-18902268273
E-mail: huh@cougarlab.org
<報道に関すること>
東京女子医科大学 広報室
〒162-8666 東京都新宿区河田町8-1
Tel:03-3353-8111 Fax:03-3353-6793
E-mail: kouhou.bm@twmu.ac.jp
CRMP1の上流には神経回路形成を制御するセマフォリン3A (Sema3A)が位置します。またCRMP1はいくつかのリン酸化酵素によりリン酸化されて、神経突起伸長やシナプス形成などの機能を果たします。今後はCRMP1が担うSema3Aの情報伝達経路においてCRMP1のミスセンスが、リン酸化修飾も含めどのような影響を与えるかを検討していきます。さらに多量体形成によりCRMP1の機能が回復するのであれば、ミスセンス変異CRMP1の複合体形成を促す化合物の探索といった取り組みも今後検討していきます。
【お問い合わせ先】
<研究に関すること>
中村 史雄(ナカムラ フミオ)
東京女子医科大学 医学部生化学分野 教授
〒162-8666 東京都新宿区河田町8-1
Tel&Fax:03-3353-8112 (Ext: 31525) 直通 03-5269-7415
E-mail: nakamura.fumio@twmu.ac.jp
Angela M Kaindl, MD PhD, Professor
Department of Pediatric Neurology & Institute of Cell Biology and Neurobiology
CharitéCrossOver (CCO), Virchowweg 6
Charité - Universitätsmedizin Berlin
Berlin, Germany
Phone: +49-(0)30 450 528429
E-mail: angela.kaindl@charite.de
Hao Hu, PhD, Professor
Laboratory of Medical Systems Biology, Guangzhou Women and Children's Medical Center, Guangzhou Medical University
School of Medicine, South China University of Technology
9 Jinsui Road, Guangzhou, Guangdong, 510623, P.R.China
Phone: +86-18902268273
E-mail: huh@cougarlab.org
<報道に関すること>
東京女子医科大学 広報室
〒162-8666 東京都新宿区河田町8-1
Tel:03-3353-8111 Fax:03-3353-6793
E-mail: kouhou.bm@twmu.ac.jp
【プレス情報】
1.掲載誌名:eLife
2.論文タイトル:Monoallelic CRMP1 gene variants cause neurodevelopmental disorder
3.著者名:Ethiraj Ravindran, Nobuto Arashiki, Lena-Luise Becker, Kohtaro Takizawa, Jonathan Lévy, Thomas Rambaud, Konstantin L. Makridis, Yoshio Goshima, Na Li, Maaike Vreeburg, Bénédicte Demeer, Achim Dickmanns, Alexander P.A Stegmann, Hao Hu,* Fumio Nakamura,* Angela M. Kaindl *
(*はcorresponding author、アンダーラインは本学所属の著者)
4.DOIコード:https://doi.org/10.7554/eLife.80793
5.論文のオンライン掲載日と報道解禁日(Embargo):13 December 2022
【English】
Monoallelic CRMP1 gene variants cause neurodevelopmental disorder
| Point ○ Researchers at the Charité - University Medicine Berlin, Guangzhou Medical University, and Tokyo Women’s Medical University found that the de novo missense variations of neuronally expressing CRMP1 cause neurodevelopmental disorders. 1.Missense variations of CRMP1 have been identified in three neurodevelopmental patients. 2.The variations occurred in the highly conserved amino acids in CRMP1. 3.The variations disrupted the oligomer formation of CRMP1. 4.Overexpression of the CRMP1 variants in cultured neurons suppressed neurite outgrowth. |
Ⅰ Background
It has been suggested that the variations of the genes involving in neural development could cause neurodevelopmental and/or psychiatric disorders using model animals including gene-targeted mice. However, the relation of human neurodevelopmental diseases and the variation of monoallelic gene is largely unknown. We, the members of Department of Biochemistry in Tokyo Women’s Medical University, have revealed that the functions of various molecules involving in neural development using primary-cultured neurons and knockout animals. We have recently reported that the tyrosine phosphorylation of Collapsin Response Mediator Protein 1 (CRMP1) regulates the dendritic development of cortical neurons (Kawashima T et al, J Neurochem 2021). Here, the group of researchers including Charité - University Medicine Berlin, Guangzhou Medical University, and Tokyo Women’s Medical University, identified the de novo variations of CRMP1 in the patients of neurodevelopmental disorder and found that the variations affect the neurite outgrowth of cortical pyramidal neurons.
Ⅱ Findings
Next-Generation-Sequence analysis of three unrelated neurodevelopmental patients identified de novo variations of CRMP1 gene. These were missense variations, i.e., the substitution of amino acid residue in the coding region of CRMP1 gene. (Fig.1).
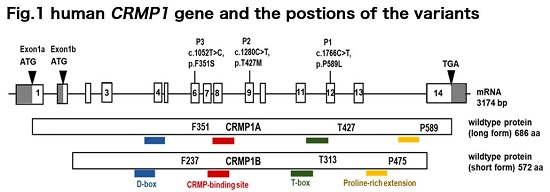
CRMP1 is predominantly expressed in the nervous system. The protein is involved in the neurite outgrowth and synapse formation during the developmental stages. It has been shown that CRMP1 knockout mice exhibited memory impairment and schizophrenia-associated behaviors. However, the relation of missense variation of CRMP1 and neurodevelopmental disorder has yet been reported.
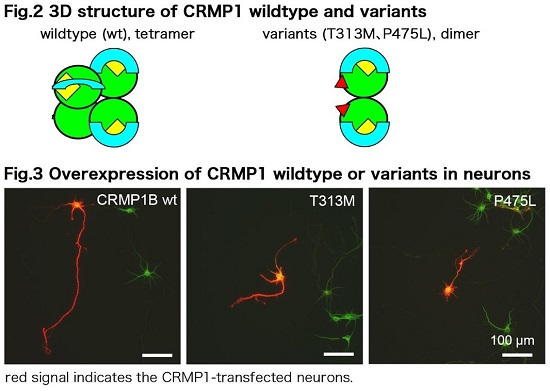
The variations in the three patients were the missense of different amino acids in CRMP1, however, each residue was positioned in the highly conserved region. It has been shown that CRMP1 protein forms homo-tetramer (Fig.2, wildtype). The structural analysis of the variants based on the known CRMP1 X-ray crystal data suggested that the missense variations would affect the tertiary structure of CRMP1. We then prepared the recombinant CRMP1 protein of wildtype and the variants and examined the gel-filtration analysis. The assay revealed that the CRMP1 variants failed to form homo-tetramer (Fig.2, variants). In addition, the overexpression of the CRMP1 variants perturbed the neurite outgrowth of primary cultured cortical neurons (Fig.3). These results suggest that the missense variations of CRMP1 may interrupt homo-tetramer formation in turn to affect the neural circuit formation and/or maturation.
The variations in the three patients were the missense of different amino acids in CRMP1, however, each residue was positioned in the highly conserved region. It has been shown that CRMP1 protein forms homo-tetramer (Fig.2, wildtype). The structural analysis of the variants based on the known CRMP1 X-ray crystal data suggested that the missense variations would affect the tertiary structure of CRMP1. We then prepared the recombinant CRMP1 protein of wildtype and the variants and examined the gel-filtration analysis. The assay revealed that the CRMP1 variants failed to form homo-tetramer (Fig.2, variants). In addition, the overexpression of the CRMP1 variants perturbed the neurite outgrowth of primary cultured cortical neurons (Fig.3). These results suggest that the missense variations of CRMP1 may interrupt homo-tetramer formation in turn to affect the neural circuit formation and/or maturation.
Ⅲ Future Perspective
CRMP1 is regulated by an axon-guidance molecule Semaphorin-3A (Sema3A). It has been shown that the phosphorylation of CRMP1 by several kinases including Cdk5 and Fyn regulates the function of CRMP1 in neurite outgrowth and synapse formation. The effect of CRMP1 variations on the synapse formation and/or maturation and the Sema3A-signaling will be investigated. Identifying the compounds facilitating tetramer formation of CRMP1 variants would be the challenging theme in the future.
<Research Info>
Angela M Kaindl, MD PhD, Professor
Department of Pediatric Neurology & Institute of Cell Biology and Neurobiology
CharitéCrossOver (CCO), Virchowweg 6
Charité - Universitätsmedizin Berlin
Berlin, Germany
Phone: +49-(0)30 450 528429
E-mail: angela.kaindl@charite.de
Hao Hu, PhD, Professor
Laboratory of Medical Systems Biology, Guangzhou Women and Children's Medical Center, Guangzhou Medical University
School of Medicine, South China University of Technology
9 Jinsui Road, Guangzhou, Guangdong, 510623, P.R.China
Phone: +86-18902268273
E-mail: huh@cougarlab.org
Fumio Nakamura, MD PhD, Professor and Head of Division
Department of Biochemistry
Tokyo Women’s Medical University
8-1 Kawada-cho, Shinjuku-ku, Toyko, 162-8666, Japan
Tel&Fax:+81-3-3353-8112 (Ext: 31525) +81-3-5269-7415
E-mail: nakamura.fumio@twmu.ac.jp
CRMP1 is regulated by an axon-guidance molecule Semaphorin-3A (Sema3A). It has been shown that the phosphorylation of CRMP1 by several kinases including Cdk5 and Fyn regulates the function of CRMP1 in neurite outgrowth and synapse formation. The effect of CRMP1 variations on the synapse formation and/or maturation and the Sema3A-signaling will be investigated. Identifying the compounds facilitating tetramer formation of CRMP1 variants would be the challenging theme in the future.
<Research Info>
Angela M Kaindl, MD PhD, Professor
Department of Pediatric Neurology & Institute of Cell Biology and Neurobiology
CharitéCrossOver (CCO), Virchowweg 6
Charité - Universitätsmedizin Berlin
Berlin, Germany
Phone: +49-(0)30 450 528429
E-mail: angela.kaindl@charite.de
Hao Hu, PhD, Professor
Laboratory of Medical Systems Biology, Guangzhou Women and Children's Medical Center, Guangzhou Medical University
School of Medicine, South China University of Technology
9 Jinsui Road, Guangzhou, Guangdong, 510623, P.R.China
Phone: +86-18902268273
E-mail: huh@cougarlab.org
Fumio Nakamura, MD PhD, Professor and Head of Division
Department of Biochemistry
Tokyo Women’s Medical University
8-1 Kawada-cho, Shinjuku-ku, Toyko, 162-8666, Japan
Tel&Fax:+81-3-3353-8112 (Ext: 31525) +81-3-5269-7415
E-mail: nakamura.fumio@twmu.ac.jp
【Article information】
1.eLife
2.Monoallelic CRMP1 gene variants cause neurodevelopmental disorder
3.Ethiraj Ravindran, Nobuto Arashiki, Lena-Luise Becker, Kohtaro Takizawa, Jonathan Lévy, Thomas Rambaud, Konstantin L. Makridis, Yoshio Goshima, Na Li, Maaike Vreeburg, Bénédicte Demeer, Achim Dickmanns, Alexander P.A Stegmann, Hao Hu,* Fumio Nakamura,* Angela M. Kaindl *
(*, corresponding authors; underline, TWMU’s authors)
4. DOI: https://doi.org/10.7554/eLife.80793
5. 13 December 2022


